CONHECENDO A BIODESTOCA
DESENVOLVIMENTO DO SETOR FLORESTAL
Nos últimos anos, a silvicultura tem se expandido significativamente no Brasil, com destaque para as monoculturas de Eucalipto voltadas à produção de celulose. A rotação dessas plantações é relativamente curta, durando, em média, até 7 anos. Após dois ou mais ciclos consecutivos de cultivo, torna-se necessária a realização da destoca procedimento que visa remover tocos e raízes remanescentes, os quais dificultam o preparo do solo e o replantio. Atualmente, o método mais comum de destoca é o mecânico (Figura 1).

PROBLEMA SILVICULTURAL
No entanto, a destoca mecânica apresenta elevado custo e está associada a diversos impactos ambientais, como compactação do solo, redução da biodiversidade edáfica e desgaste prematuro de equipamentos e ergonomia dos recursos humanos.
Na busca de alternativas que visam contornar esses problemas, estudos vem sendo realizados para preencher essa lacuna no setor florestal, como o uso de microorganismos xilófagos para acelerar a biodegradação e facilitar ou mesmo substituir a destoca mecânica.
Hipótese principal e objetivo do projeto
Hipótese: Enzimas microbianas são capazes de acelerar a degração dos tocos remanescentes facilitando o preparo do solo para novos plantios.
Objetivo principal: Desenvolver um acelerador de decomposição biológico, formulado com enzimas lignocelulolíticas, agentes estabilizantes e microrganismos benéficos. Uma alternativa viável, ecologicamente correta e altamente promissora para acelerar o processo de biodegradação de tocos remanescentes no campo.
Perguntas e respostas
Quais microorganismos são produtores potenciais?
A escolha dos microorganismos deve se basear em alguns requisitos já estabelecidos na literatura:
devem ser fungos da podridão da madeira (podridão branca);
não patogênicos;
encontrados no subbosque de reflorestamentos.
Objetivo: Encontrar fungos xilófagos com potencial enzimático para serem aplicados na biodegradação acelerada de tocos remanescentes em reflorestamentos comerciais.
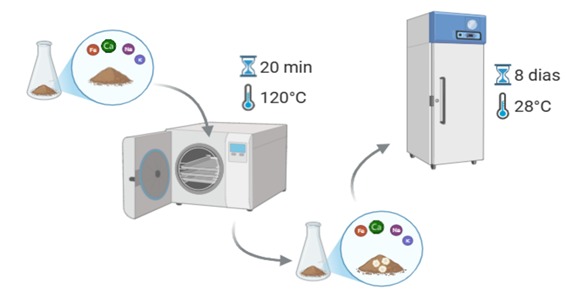
1° passo: Coleta de materiais em reflorestamento
As coletas de madeira com sintomas de podridão e estruturas fúngicas foram realizadas em plantios de Pinus e Eucalipto em fazendas comerciais da empresa CMPC, nos estados do Rio Grande do Sul e Paraná. (Figura 2).

2° passo: Isolamento e coleção de culturas
Os materiais foram encaminhados ao Laboratório de Patologia Florestal, onde foi realizado o isolamento direto e indireto de diversos fungos, e culturas puras foram obtidas para criação da coleção de isolados fungicos a serem testados.
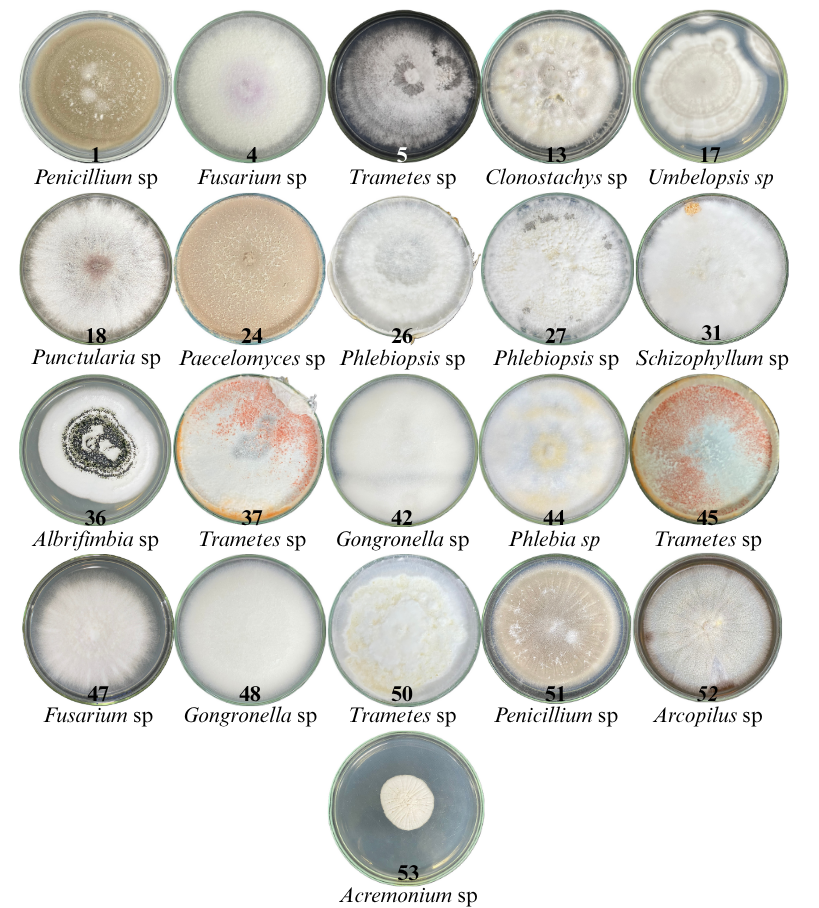
Resultado: Coleção de isolados CMPC
Foram obtidos 53 isolados fúngicos dos materiais coletados no Sul do Brasil.
Quanta diversidade, não é?!
3° passo: Triagem de perfis enzimáticos
Com a obtenção da coleção de fungos, a próxima etapa é a verificação das atividades enzimáticas de cada isolado para conhecer quais seriam produtores potenciais para aplicação. Para isso foram estabelecidas as enzimas de interesse, baseando-se nas enzimas protagosnistas na degradação dos componentes da parede celular vegetal (celulose, hemicelulose e lignina). São elas:
Xilanase;
Endoglucanase;
FPase;
Lacase;
β glicosidase;
β xilosidase.
Metodologia: Fermentação semissólida e Extração de Enzimas
Para isso, foram utilizados três substratos diferentes: Casca de soja, Farinha de trigo, Serragem de Pinus.

Etapa 1: Indução dos isolados nos substratos
Etapa 2: Extração das enzimas
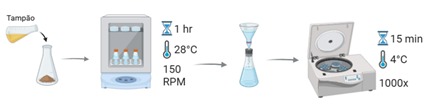
Para medir a atividade de enzimas utilizou-se o método colorimétrico de Miller.
Para cada isolado foram realizadas 2 repetições com 3 réplicas em cada uma das biomassas.
Resultados: Melhores produtores de enzimas lignoceluloliticas
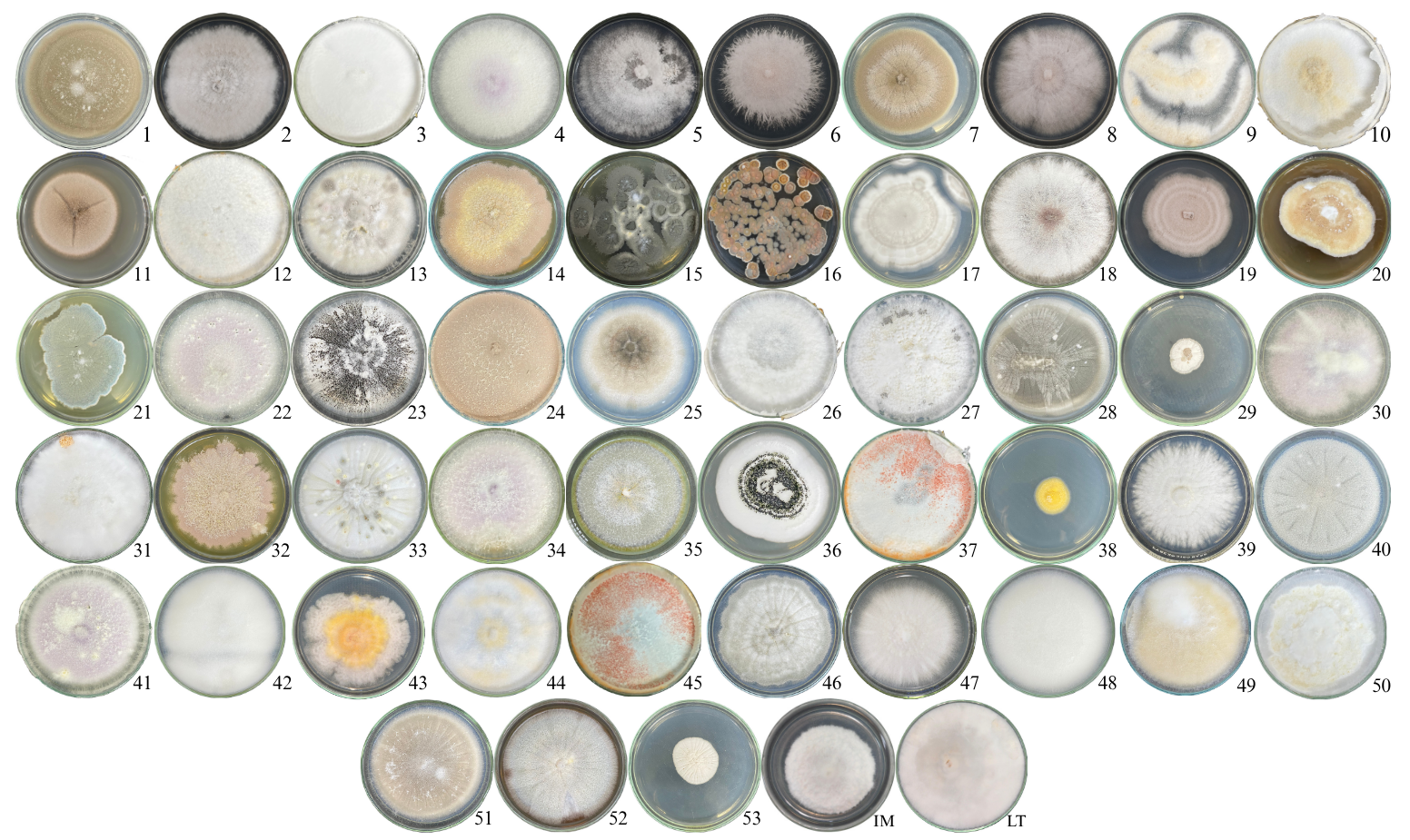
A partir das atividades enzimáticas, foram selecionados 21 isolados que apresentaram as melhores produções das enzimas de interesse, essa seleção, determinou a melhor biomassa indutora para cada organismo selecionado. Você pode conferir a estatistica de seleção, clicando aqui.
4° passo: Identificação a nivel de gênero dos isolados selecionados
Os isolados mais atrativos foram identificados através de PCR (Reação em Cadeia da Polimerase) e Sequenciamento de DNA. As sequências obtidas foram comparadas com bancos de dados para identificação dos gêneros.
Metodologia: Extração de DNA e PCR
Para extrair o DNA genômico total dos isolados, foi utilizado o kit de extração de DNA de fungos Wizard Promega, posteriormente amplificado pela reação em cadeia polimerase (PCR) de acordo com o protocolo fornecido pelo fabricante. A região ITS foi amplificada utilizando o par de primer ITS1/ITS4 (White et al.,1990), para casos específicos o par de primer ITS5/ITS4 foi utilizado, utilizando o seguinte protocolo:
desnaturação inicial a 96°C durante 5 minutos;
35 ciclos a 94°C por 30 segundos, 50°C por 30 segundos e 72°C por 1:30 minutos;
extensão final a 72°C durante 4 minutos.
O sequenciamento de DNA foi realizado e o processamento das sequências foi feito no programa SeqAssem, para obtenção da sequência contig, e a partir do SeqAssem, foi utilizada a ferramenta de alinhamento Blast, do Banco NCBI (ALTSCHUL et al., 1980). Sequências acima de 98% de identidade foram consideradas comoespécies equivalentes.
Resultado: Biodiversidade de Gêneros
Dentre os 14 gêneros identificados, predominam os ascomicetos com 9 isolados e 7 gêneros seguidos dos basidiomicetos com 9 isolados e 5 gêneros, sendo que a ordem Poliporales dentro do filo Basidiomycota apresenta maior frequência de isolados, isso porque o gênero Trametes sp, apresentou maior frequência de isolados.